近年来,长读长 PacBio HiFi 测序为基因组学的许多领域做出了变革性的贡献,包括 <17>De Novo</17> 组装、所有变异类型识别、全长定相靶向和全基因组测序、全长 RNA 测序和高分辨率宏基因组学。 做出这些贡献的基础是对天然长 DNA 分子进行高度准确的端到端测序。 近十年来,研究人员多次尝试利用短读长技术从短序列 reads 中合成重建较长的分子序列,他们通常会在此过程中使用 PCR 来复制原分子。 事实证明,使用与 Moleculo 或 10X Genomics 关联的 reads 进行样品制备和分析非常麻烦,因此,研究人员后来便不再使用此类 reads。 最近,Illumina 宣布推出“Infinity”reads,而 Element Biosciences 则收购了 Loop Genomics,更新了合成长读长的概念。
尽管目前很少有关于这两种新的合成长读长测序方法的公开数据,但 Illumina 于今年早些时候在基因组学与生物数据节会议 (FoG 2022) 上展示了一项实例比较。 在下方的 IGV 屏幕截图中(下图),合成 Infinity reads(标记为“Longas”)位于顶部,其后是标准 Illumina 短读长,而被标记为“CCS”的 PacBio HiFi reads 则位于底部:
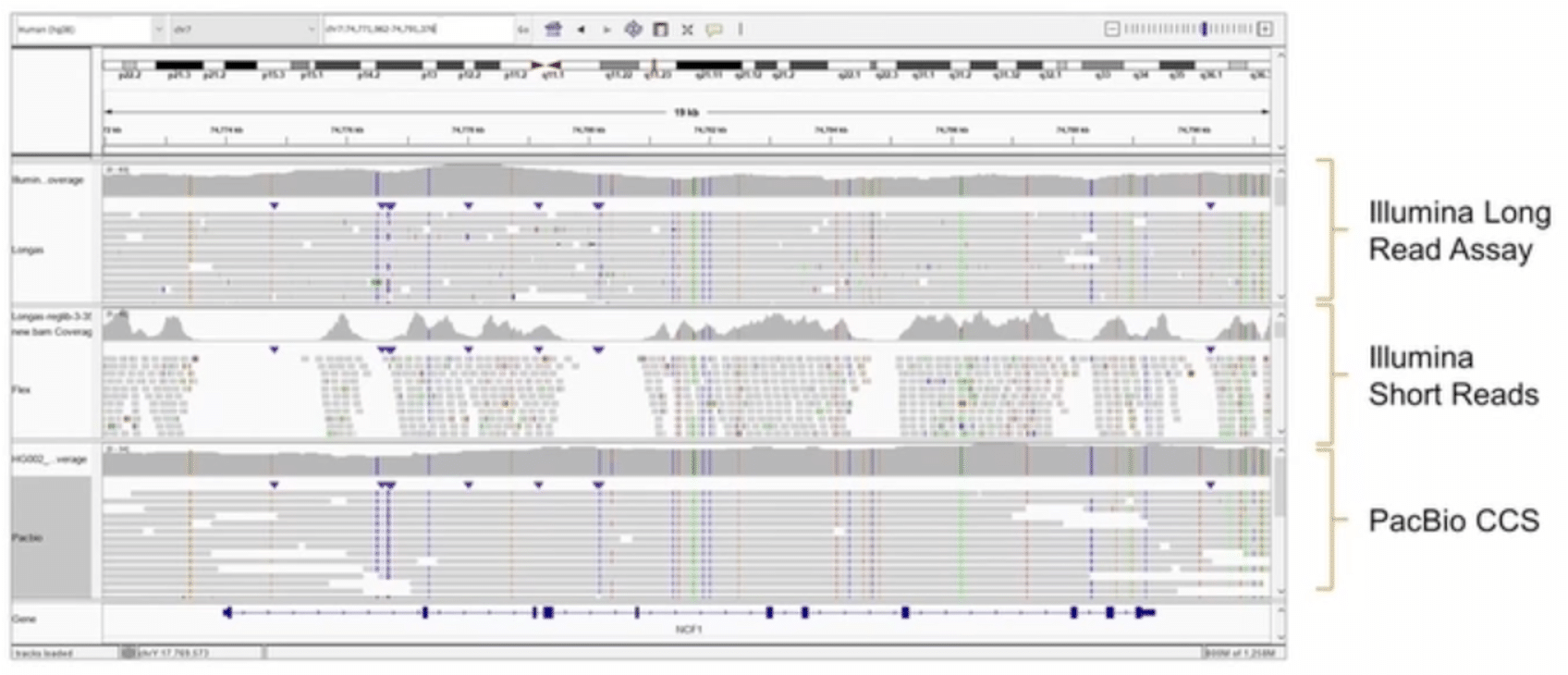
但是,如果进行仔细比较,就会发现 PacBio HiFi reads 具有重要的优势(图 2),包括:
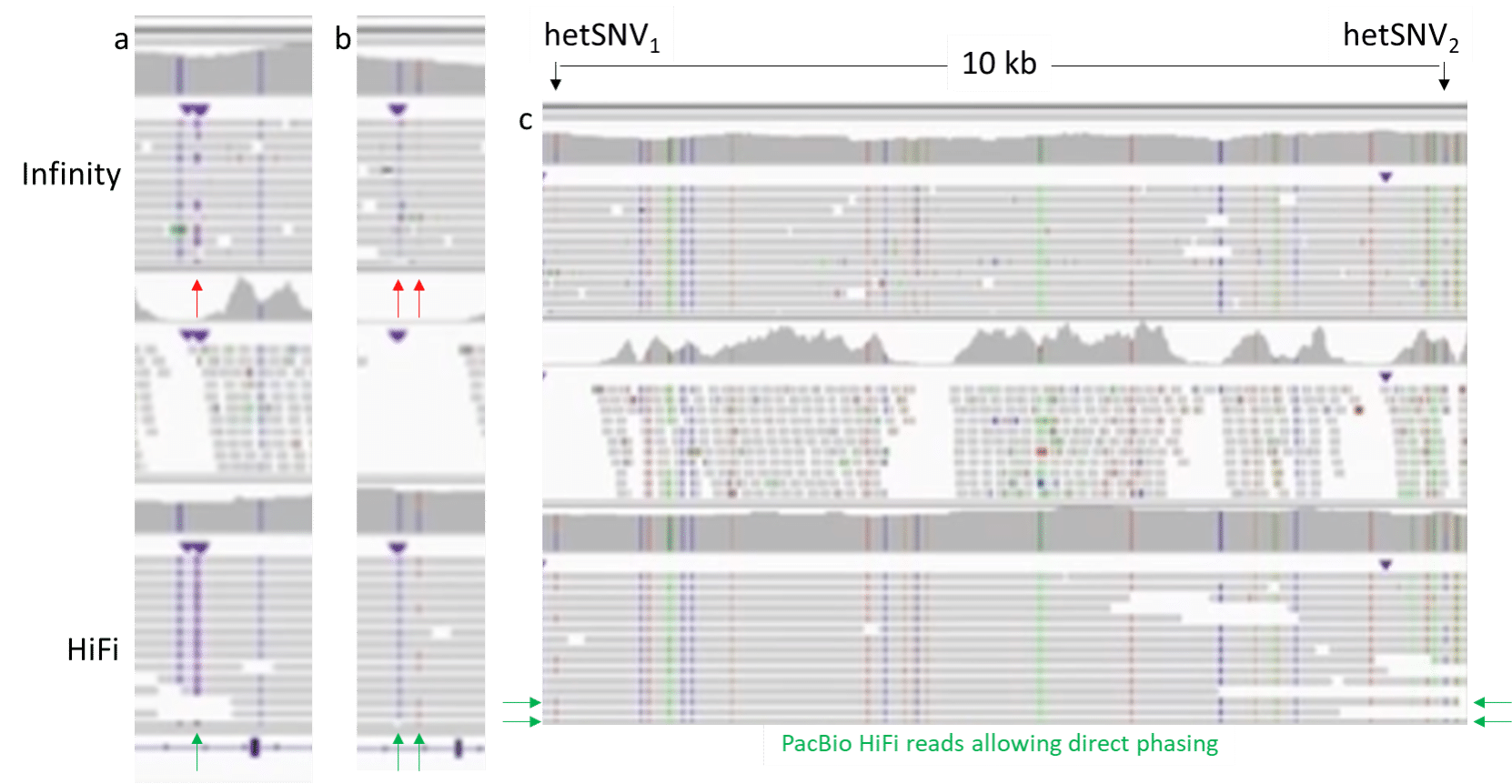
a) 变异检出的准确性: 对于 PacBio HiFi reads 明确解析出的一个纯合变异(串联重复插入),Infinity 仅检测到了该变异的一部分,并且将其错误地显示为杂合变异。
b) 变异检出的置信度: 对于 PacBio HiFi 明确解析出的两个相邻纯合和杂合 SNV 的区域,Illumina Infinity 覆盖度 panel 也检出了该变异,但底层 reads 数据支持不足。
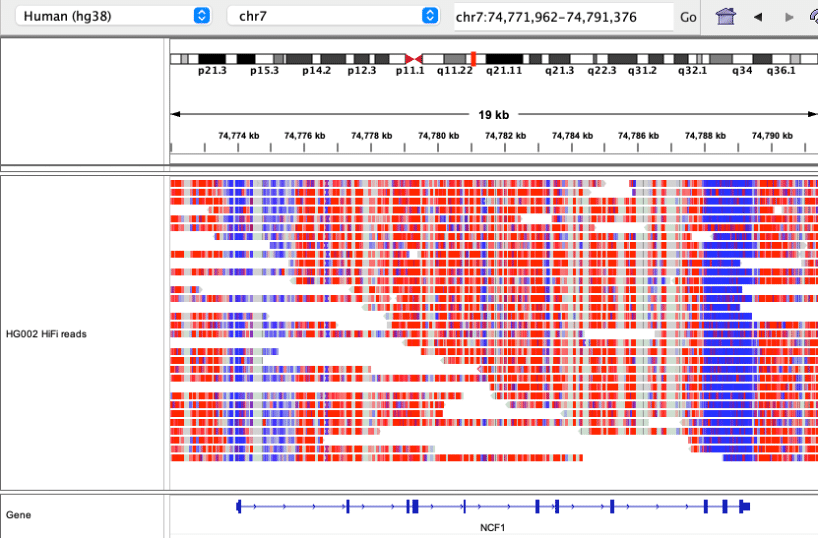
c) c) 长距离定相: 上述 IGV 区域包含杂合变异,间隔约 10 kb,通过完全跨越该距离的几个 PacBio HiFi reads 明确定相,进而实现了直接定相(相对于参考样本,为两个变异的顺式排列)。 Illumina Infinity reads 长度不够,无法跨越两个变异之间的距离。
此外,真正的长读长测序能够直接检测 CpG 甲基化,而短读长测序则无法实现,用于各种合成 reads 方法的 PCR 也是如此。
本周,Element Biosciences 描述了使用 Loop Genomics 技术的合成长 reads,并展示了宏基因组扩增子测序的技术比较图(图片引用自 Callahan et al. (2021) 论文中的图 3):
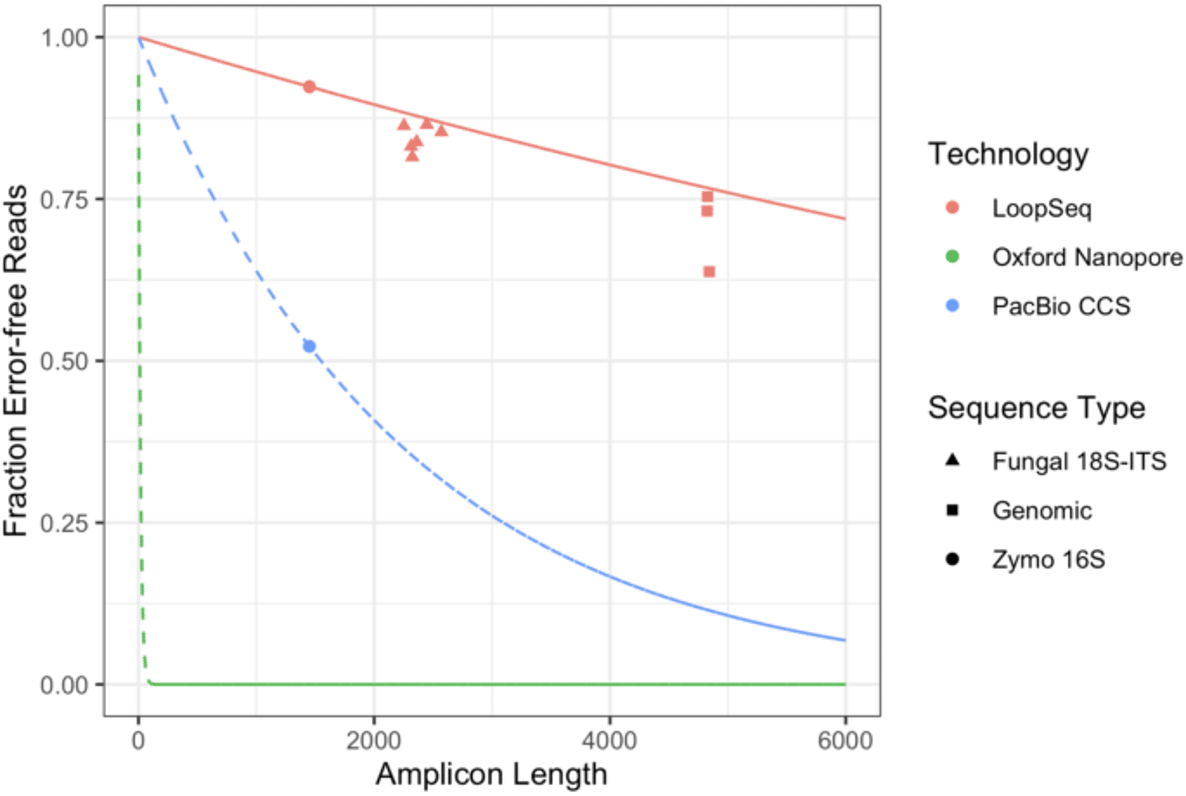
“PacBio CCS”数据点取自 2019 年发布的一篇论文,不代表当下性能。 例如,Karst et al. (2021) 以 99.9993% (Q51.5) 的准确度描述了同一样本的全长 (4.4 kb) rDNA 扩增子的 HiFi 测序,几乎比之前的研究高出了两个数量级。 将此数据点添加至图表中后,HiFi 测序的卓越准确性便得以展现,同时也为我们解答了 HiFi 测序将如何实现更好的应用性能:
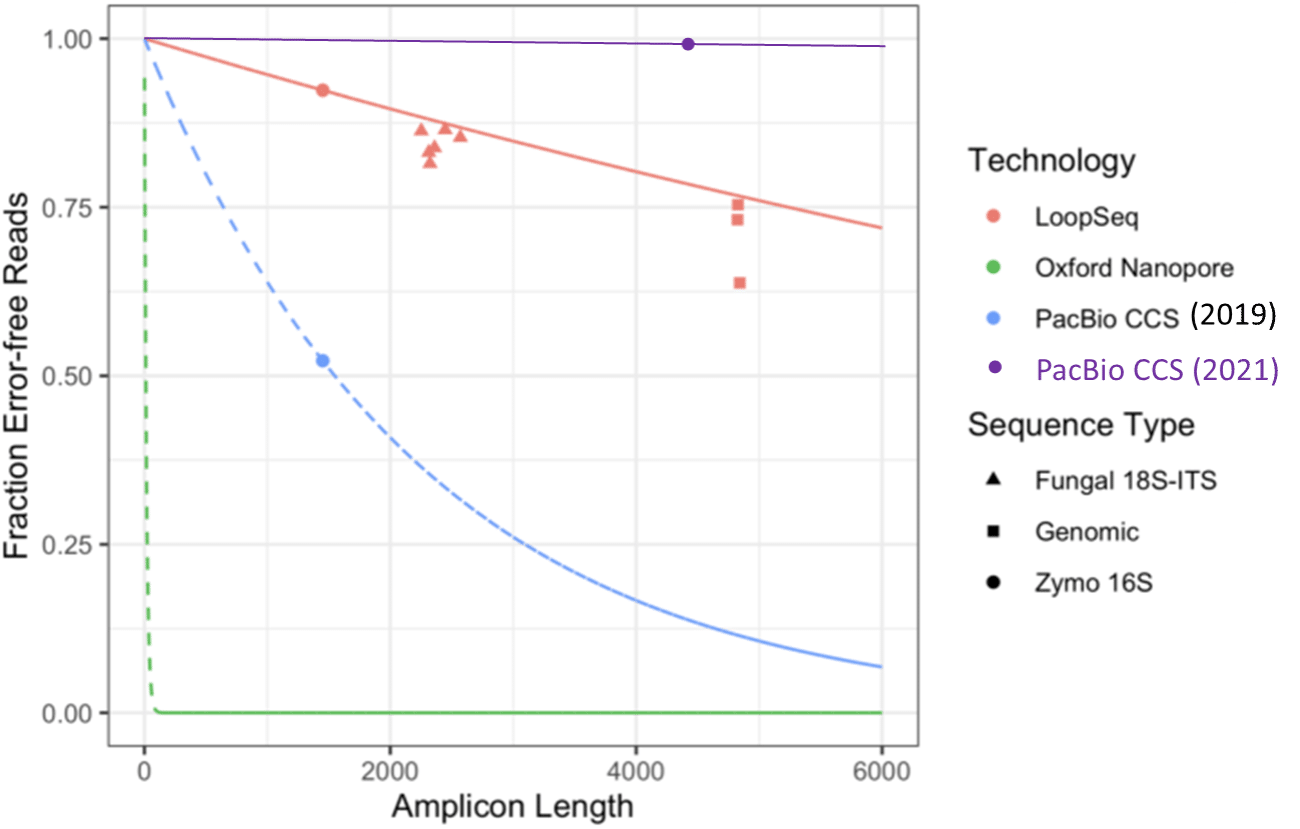
这些示例再次印证了科学界多年来得出的结论——合成长读长测序根本无法与 PacBio HiFi 测序产生的真正长读长测序的优势相提并论。 此外,合成长读长测序还存在提供不准确信息的可能,进而产生的杂峰则会干扰研究。
PacBio HiFi 测序能够提供最准确、最连续、最完整的基因组和表观基因组信息,得到了越来越广泛的应用,助力基因组学履行改善人类健康的承诺。
