上周,端粒到端粒 (T2T) 联盟宣布,他们获得了人类基因组的第一个完整序列。时至今日,科学界依然对这项惊人的成就议论纷纷。 得益于长读长测序技术的进步,以高度完整性、连续性和准确性从头到脚组装基因组的目标已经实现。 事实上,在《Nature》发布的今年有望震撼科学界的年度技术中,完整基因组被列入“2022 年值得关注的技术”,位居第一。
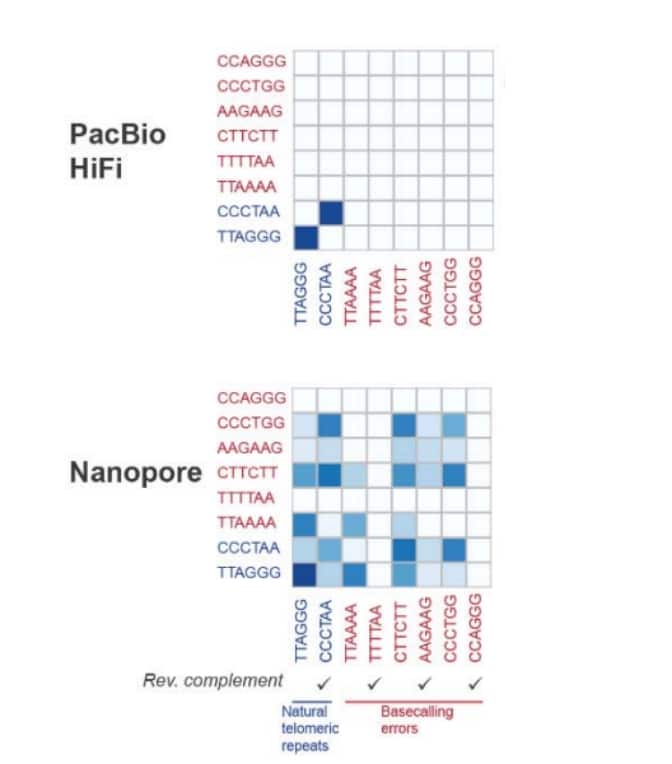
若想生成 T2T 的基因组,我们不仅需要具备对端粒进行测序的能力,同时也需具备其他能力。 其他学者也报告过类似的发现。 Chris Mason (Weill Cornell) 博士及其合作者最近在 Genome Research 上发表的一篇文章强调了 PacBio 提供的准确、长 HiFi reads 在解析二倍体基因组中的两个端粒等位基因方面的能力,观察了人类端粒中新的单倍型多样性和序列异质性。
除了人类基因组测序以外,研究人员目前也能够在植物和动物基因组 HiFi 组装中经常观察到完全组装的端粒。 例如,Yu et al. 在近期发布的一篇预印本论文中使用 PacBio 端粒测序发现了第一个与端粒酶无关的脊索动物体内末端复制问题解决方案。 具有高度再生能力的欧非肋突螈缺乏端粒酶活性,其基因中含有在序列和结构上与所有已知脊椎动物都不同的端粒,而且,这种蝾螈利用一种端粒延长替代 (ALT) 机制来维持生理性端粒。 此外,研究人员还在人类癌细胞系子集中观察到了 ALT 可以补偿的端粒损耗。
鉴于生成完整基因组的重要性和它们在衰老或肿瘤等领域内的生物学意义,对端粒和基因组的其余部分进行准确且完整测序的能力至关重要。 PacBio HiFi 测序是研究人员的信心之选,每一名研究人员都坚信他们的数据将成为研究道路上最坚实的基础。
