PacBio HiFi 测序可提供更为精确的数据,能够显著影响遗传关联性研究的分辨率,减少所需的队列规模。
例如,近期,费城儿童医院的研究人员及其合作者发表了一篇题为“ 经过改进的进化选择检测方法突出了针对复杂基因组区域的不同测序策略的潜在偏倚” 的预印本论文,描述了平衡选择之下运用 HiFi 技术检测基因组区域的情况。
研究人员在 497 个基于芯片和 NGS 外显子组的临床数据集、来自 IHIW 数据库的 3500 条基于 NGS 的 HLA 分型数据以及来自人类泛基因组参考联盟 (HPRC) 的 23 个高质量 PacBio HiFi 基因组中测量了关联性信号。 尽管 PacBio 数据集的队列规模缩减十倍以上,但全基因组连锁不平衡 (LD) 分析以及关注 MHC 区域的图表突显出在信噪比方面存在的巨大差异:
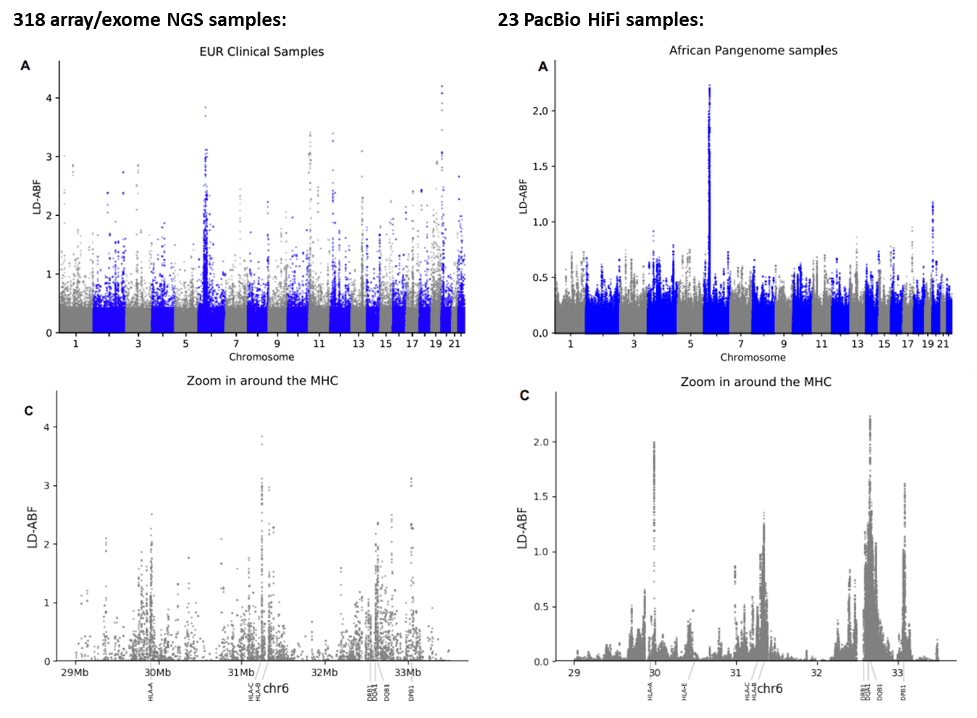
PacBio HiFi 队列分析从 NGS 队列分析中发现了几个错误信号,这非常重要。 作者写道:
“很明显,在非洲泛基因组样本中,我们在临床样本能看到的 SIRPB1 信号并不存在,这表明 它们有可能是不准确的【短读长】序列定位导致的杂峰”。 此外,我们还在 MHC 区域内观察到另一个来自短读长 NGS 分析的假阳性信号: “在 IHIW 数据观察到的集中在 [HLA] -DRB1 的内含子 5 的显著峰值在泛基因组分析中完全不存在。 已知这部分 DRB1 具有 结构变异和重复元素,这阻碍了较短测序 reads 的准确定位,因此可能导致 IHIW 中出现杂峰 LD,而泛基因组中则未出现。”
作者得出结论:
“我们得到的结果表明,与来自其他平台的较大测序数据集相比,数量级较小的高质量长读长测序数据集也许对于表征遗传变异而言更为有效。”
PacBio HiFi 测序提供的序列信息更加精确,进而帮助我们更好地理解遗传多样性,以及理解对于不同个体而言“正常”生物标志物水平的判定。近期,由美国国家过敏和传染病研究所 (NIH) 的研究人员与来自 28 家参与机构的合作者共同发布的一篇预印本论文,可作为该观点的有力佐证,论文题为“ 在遗传方面确定生物标志物类胰蛋白酶的个体化临床参考范围能够减少不必要的程序并能反映髓系肿瘤”。 在这项研究中,PacBio 测序被用于解析人类基因组中编码酶(血清类胰蛋白酶)的复杂区域。血清类胰蛋白酶的水平升高是某些髓系肿瘤的生物标志物。 研究人员通过观察发现,在一些人的体内,该位点中的一个基因 TPSAB1 在 15 kb 串联重复中被复制。
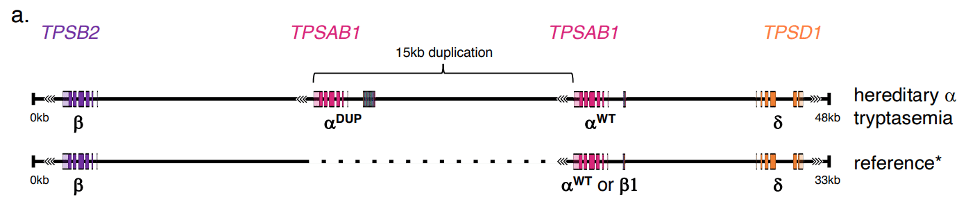
此外,他们还识别出一系列独特的近端非编码变异,能够将复制序列与非复制序列区别开来。 5′-UTR 之内的扩展 DNA 基序也与 <110>TPSAB1</110> 复制相关变异有关,其体外启动子活性大于非复制启动子中的旁系同源区域。
作者利用这些新见解,根据基因的复制数为生物标志物的参考范围创建了一个新的基于遗传的模型,从而 设定出“全新的‘正常’上限个体化临床参考值”。 因此,对于某些存在这种复制的个体而言,此前通常提示需要骨髓活检的生物标志物水平升高,如今被视为在“正常”限度内,由此便避免了费用高昂的侵入性活检。
这些研究和其他研究有力地证明了,更加全面地了解个体基因组将帮助我们更好地认识一个人的健康或疾病状况的遗传基础,从而为其提供更合理的治疗。 这也正是精准医疗的前提。 PacBio HiFi 测序能够提供更准确、更连续、更完整(即更精确)的遗传信息,是一项关键的赋能研究技术,有助于实现基因组学的承诺——通过精准医疗改善人类健康。
PacBio 产品仅供科研使用,不得用于诊断程序。
