如今,越来越多的研究人员都因 PacBio HiFi reads 的组合测序准确性和长度而受益,每周都有许多新论文和预印本论文发布。 几个月前,Illumina 宣布尝试以合成的方式通过从短读长测序中创建较长的序列信息来模仿 HiFi reads,并将其命名为 Infinity reads。
这代表了科研界对 HiFi 长读长测序在人类基因组学领域内的价值的广泛认可。仅在过去一周内,就有多项利用 HiFi reads 的研究发布,内容包括:用于 LD 研究的复杂基因组区域的分辨率有所提高、对肾病的致病变异的灵敏度有所提高、对疾病生物标志物参考范围的遗传决定更加明智、对乳腺癌样本结构变异的检测有所改进以及对人类基因组倒置的表征和人类端粒 DNA 和染色质的单分子结构。
我们由衷感谢科学界将原生长读长 PacBio HiFi 测序用于越来越多的人类基因组。如今,这种技术还可于 同时解码表观基因组。
在本周的早些时候,来自 Illumina 的 Gary Schroth 博士在一场<61>网络研讨会</61>上展示了几个示例,对 Illumina Infinity reads、标准 Illumina reads 和 PacBio HiFi reads 进行了比较。 事实上,此次比较有力地证明了 HiFi 测序数据质量的优越性,也展示了 Infinity 合成长读长测序技术所引入的多个重大错误和杂峰。
Schroth 博士以 IGV 屏幕截图的形式展示了人类基因组的几个区域,将常规 Illumina 测序与 Infinity 合成长读长数据进行了比较。在比较过程中,PacBio HiFi 测序数据被用作市场上的金标准和真实对照。
我们来仔细观察其中的两个例子。 第一个例子涉及 15 号染色体,读长达 23 kb,含有 STRC 基因(在旁注中,所有示例都清楚表明标准 Illumina 短读长测序在这些区域内表现不佳):

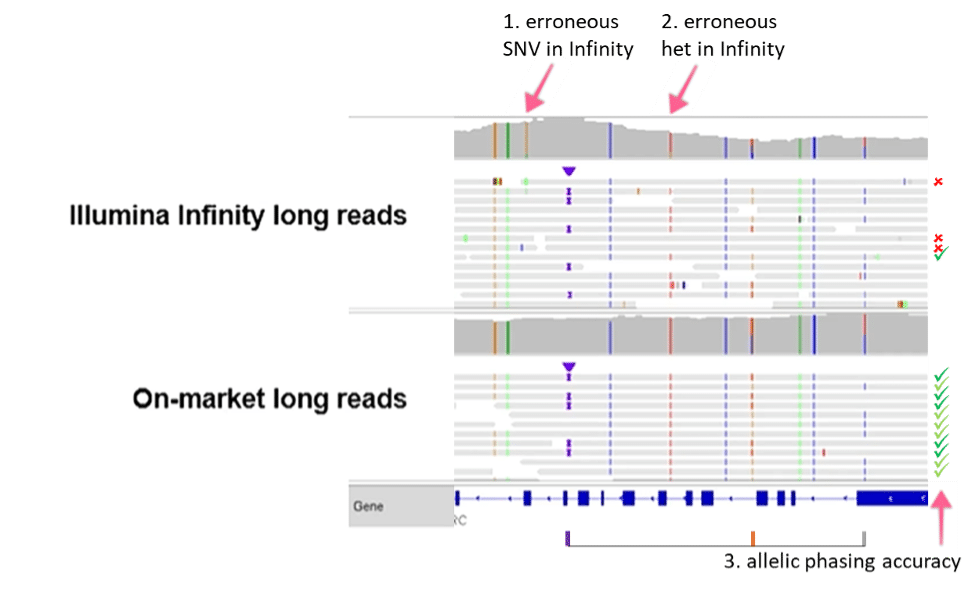
放大中心附近的部分并将 Infinity 合成长读长与 PacBio 原生长 HiFi reads 进行比较,结果显示 Infinity 数据中存在几个错误:
1. 假阳性 SNV
2. 一个纯合 SNV 被误检为杂合 SNV
3. 三个杂合变异的定相不正确。 在该示例描述的 14 个 Infinity reads 行中,10 个未提供信息,因为 Infinity reads 的长度不足以跨越该区域。 在有 reads 能够跨越该区域的其余四行中,三行给出了错误的答案。 相比之下,全部 11 个 PacBio HiFi reads 的长度对于提供信息而言都足够充分,而且,11 个 reads 均提供了两个等位基因的正确相位信息,清晰地显示了前两个顺式变异和第三个反式变异。
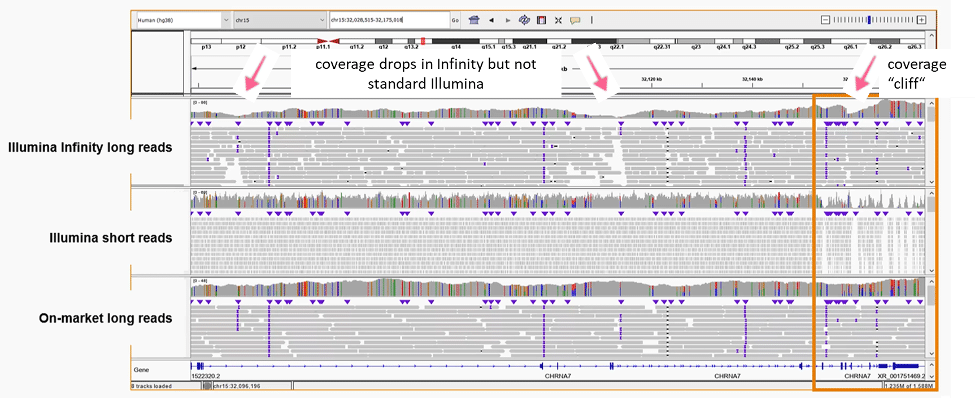
在第二个例子中,Schroth 博士展示了 15 号染色体上的一个 145 kb 区域,其中包含 CHRNA7 基因。 在上述区域内,我们可以看到,在 Infinity 数据中出现两次覆盖度显著下降,阻碍了这些区域的可信变异检出和定相。 实际上这两个区域均在标准 Illumina 测序的覆盖范围之内,这表明新的覆盖度缺口似乎是通过合成长读长引入的。 在位于右侧的演示文稿中以橙色突出显示的区域内,我们可以在 Infinity 数据中看到一个覆盖度“断崖”,表明存在数据质量问题。 相比之下,在他展示的所有示例中,PacBio HiFi 的覆盖度均匀,能够提供可信的变异检出和定相。
其他示例的情况与此相似。 值得注意的是,示例中还显示了曾在 1 月份展示 的一张 NCF1 基因屏幕截图,图中仍然包含先前突出显示的 该区域内的许多错误。
与合成长读长相比,原生 PacBio HiFi reads 的质量较好,原因包括:
1. 分子完整性:由于对从细胞中提取的天然 DNA 分子直接进行测序,因此与合成方法相比,PacBio HiFi read 测得的 DNA 片段要长得多。 例如,在上方的 STRC IGV 屏幕截图中,Infinity reads 的平均长度为 4.5 kb(中位数为 3.8 kb),要比人类 WGS 的典型 PacBio HiFi 文库(18-20 kb)短 4 倍以上。 准确序列 reads 的连续性较好,改善了所有变异类型的检出、单倍型定相、覆盖均匀性、5mC 检出和表观遗传定相 以及许多其他方面的结果,包括人类、植物、动物、细菌和病毒基因组和宏基因组群落的 De Novo 组装等。
2. 样品制备步骤更简单,无需扩增:根据定义,天然 DNA 分子测序不需要任何 DNA 扩增或其他会改变序列的分子生物学程序,而每个此类程序都容易出现偏差、引入错误、片段化和其他杂峰。 另一方面,合成长读长却需要完成这些繁琐的步骤。 PacBio HiFi 可直接对未经改动的 DNA 分子进行测序,不仅便于用户查看整个基因组(包括由于短读长测序仪本身的局限性而被视为难以用 NGS 测序的区域)的全貌,而且不会导致出现此类偏差,而使用合成的长读长结构则无法克服这一挑战。 此外,扩增会导致合成长读长测序中的甲基化信息完全丢失;相比之下,5mC 测序如今已成为 PacBio HiFi 测序不可或缺的一部分,减轻了用户的负担。
3. 生物信息更简单:对长天然分子进行测序还可以简化后端的生物信息。 与合成长读长测序相比,PacBio 原生长 HiFi reads 不需要生物信息来指定哪些小片段源自同一个原始长分子,也无需复杂的 reads 组装步骤、纠正样品制备过程中引入的错误和/或从对照样品中删除任何信息。 即使这些步骤在短读长测序仪上都处于隐藏状态,它们也有可能引入错误。
因此,正如我们在近十年中看到的那样,研究人员对合成长读长方法进行了无数次尝试,但均以放弃告终。长而准确的原生 PacBio HiFi reads 能够提供最准确、最连续、最完整的基因组和表观基因组信息,其应用范围越来越广,性能也远优于合成长读长测序。 此次网络研讨会也重点展示了短读长测序所提供的标准结果的完整性有多差。 正如演示内容所示,PacBio HiFi 测序现已上市,能够以一流的序列质量呈现基因组的完整视图,让研究人员对其数据和研究结果充满信心。
