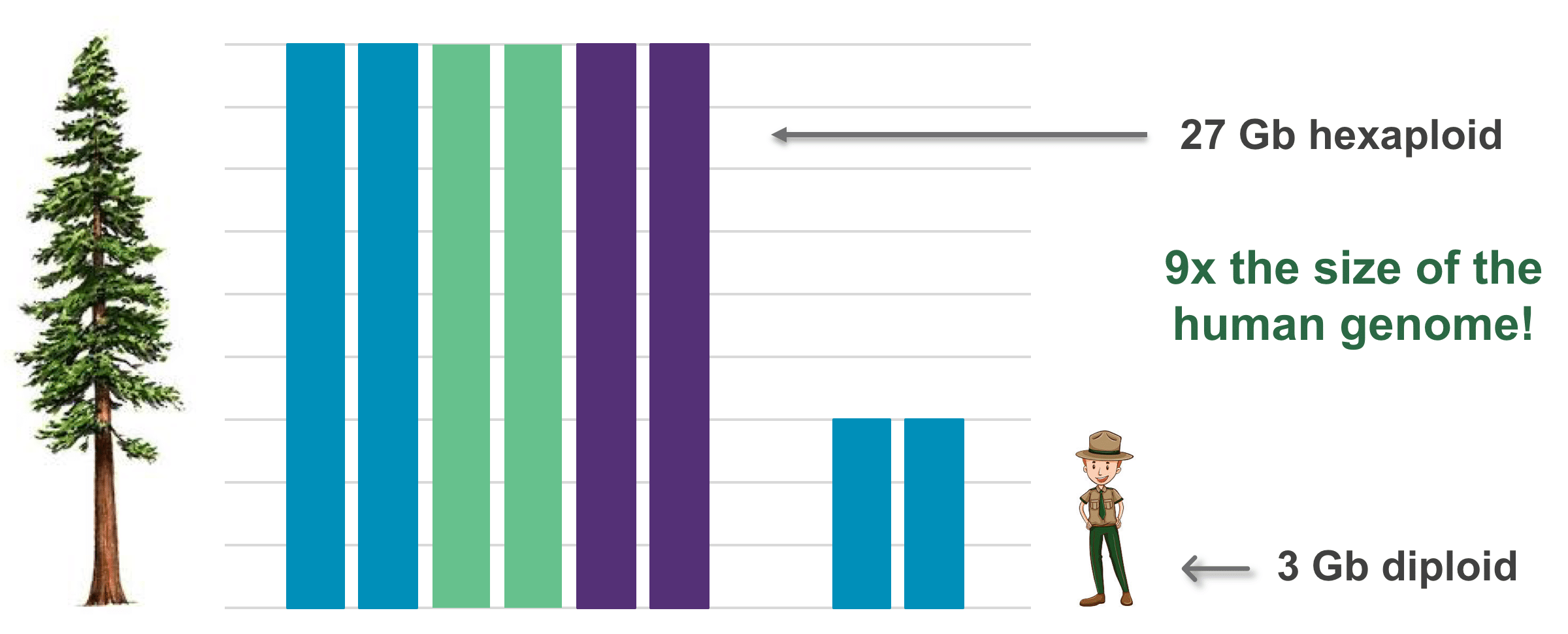
加利福尼亚红杉: 一种高度和年龄上的巨树(高达 379 英尺,树围达 29 英尺,年龄达数千岁),这种著名的高耸树木也源自于一个巨大的 27 Gb 基因组。
为了给 Sequel II 系统寻找测序挑战,我们选择了加利福尼亚红杉,也就是科学家所说的 Sequoia sempervirens. 在 Stanford University(斯坦福大学)附近刚好有不少优质的标本。
由 PacBio 科学家——Emily Hatas 、Greg Young 和 Michelle Vierra 几人组成的科研小组带着冰、剪刀和厨房秤前往校园采集了相关样本。
他们使用 Circulomics Plant Nuclei 试剂盒分离了样本的 DNA,构建了 HiFi 文库并进行了测序。 在短短 7 天内,团队便获得了基因组的 22 倍覆盖(606 Gb HiFi 数据)。
又过了 6 天,Greg Concepcion 生成了部分解析后的单倍型的基因组组装,其大小几乎是预期基因组大小的 2 倍,contig N50 为 1.92 Mb。
“结果非常令人惊讶,”Vierra 说。 “我们非常高兴看到这一基因组组装与其他最近的松柏科基因组相比所表现出的进步。”
巨大的基因组仅用了 17 天就组装完成——4 天用于样本制备,7 天用于测序,6 天用于组装,详细介绍可参见 Vierra 发表在媒体专栏中的报道。
但团队的目标不止于此。
作为一般推荐,10 至 15 倍覆盖度的 HiFi reads 是获得基因组的理想测量范围,有利于 3 C 基因组质量的测量。
对于加利福尼亚红杉大小的基因组,在有限的时间内获得如此高的覆盖度可能不经济也不可行,因此团队倾向于了解合理的约 20 倍的覆盖度能产生什么。 在多占用了一些时间且有足够的 HiFi 文库支持的情况下,团队成员继续测序,获得了总共 875 Gb 的 HiFi 数据,代表对基因组 33 倍的覆盖。
结果不出所料,好材料 (HiFi reads) 越多,基因组组装效果就越好。 连续性显著改善,contig N50 为 3.8 Mb,完成度增加,完成 BUSCO 评分接近 61%。
总之,几年前还被视为巨量的工作被几个人在业余时间仅用了几周就完成了。 我们希望,随着 PacBio HiFi reads 使用的不断增加,我们将继续看到全部基因组组装的改进,包括巨大以及更加复杂的多倍体植物。
红杉基因组的更多数据和更新的基因组组装现已公开,可点击此处获取。 我们希望它将成为所有松柏科研究人员的实用工具!

加利福尼亚红杉基因组组装结果的比较。 [1] Hybrid assembly of redwood. [2] Transcript set of Abies alba from Neale et al. Varying number of transcripts aligned to each genome (4,958 mapped to 22-fold HiFi Reads, 4,970 mapped to 33-fold HiFi reads, 4,760 mapped to ONT) [3] Assembly with 33-fold HiFi reads was done with 80 cores and an updated version of Hifiasm (0.3.0).
—
您是否有兴趣了解更多关于测序您的目标生物所用 HiFi 数据的相关信息? 联系 PacBio 科学家 讨论您的项目需求。
