 无论是增强标记开发、促进生产更有营养的食品、保护牲畜健康还是提高作物产量,从全面 HiFi 测序中获得的生物学见解对于农业行业来说都是不可估量的。
无论是增强标记开发、促进生产更有营养的食品、保护牲畜健康还是提高作物产量,从全面 HiFi 测序中获得的生物学见解对于农业行业来说都是不可估量的。
继 AGBT AG 三部分系列中的第一部分之后,我们将探讨 PacBio 如何提供前沿测序解决方案,使农业科学家们能够以可持续地方式满足日益增长的人口需求。
新一代测序工具
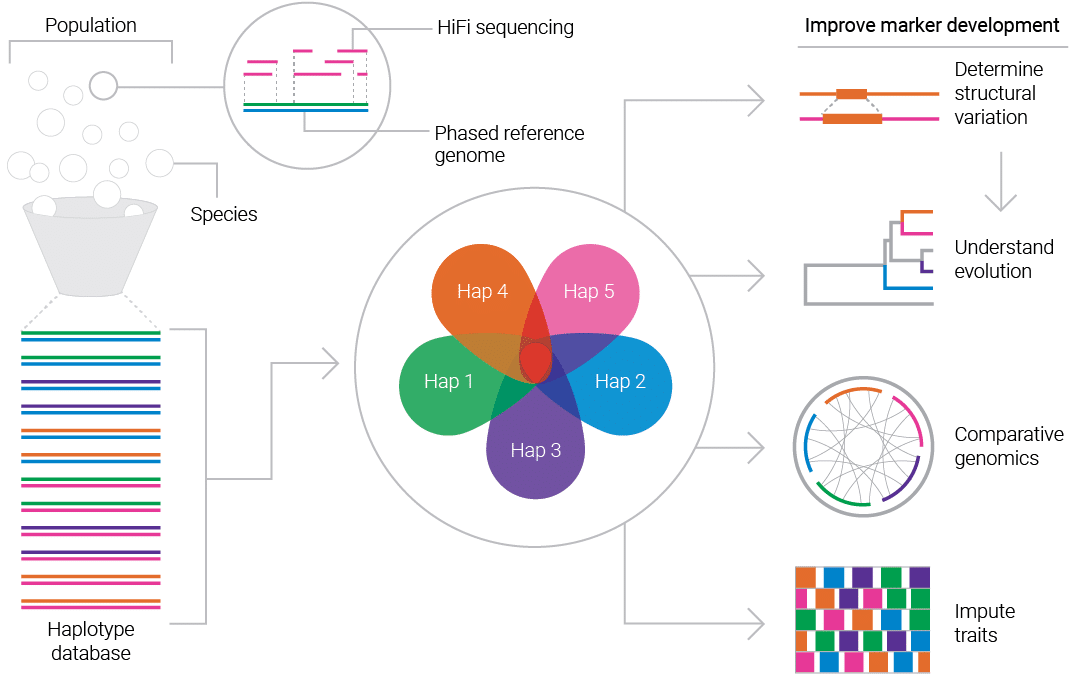
全基因组测序为育种家提供了数据驱动型策略,通过识别理想性状进行分子标记辅助育种。 然而,动植物生物学比较复杂,这些性状往往也非常复杂,无法归因为单个 SNP, 需要在完整的单倍型中进行理解。
因为 HiFi 测序本质上为单分子测序,准确度高,读长可达数千个碱基,因此理想情况下,即使是十分复杂的动植基因组也可以快速组装定相的参考级别质量的基因组。
借助 HiFi 测序,科学家们能够:
- 利用 SNP、结构变异和复杂基因型实现理想性状
- 在全基因组范围内捕获远交系、近交系和种群中的基因组变异
- 构建参考级别质量的单倍体分型的泛基因组,以推动标志物的开发、性状发现和种质表征
水果育种中的苹果品种培育
大多数作物具有复杂的基因组,其特征是尺寸大、杂合性水平高且为多倍体。
从极具挑战性的 2.3 Gb 玉米(其中 85% 由高度重复的转座因子组成)到节段异源四倍体玫瑰和大豆(其拥有惊人的 60,000 个适应不同生态区域的种质),HiFi 测序不仅帮助科学家们为更多复杂个体系构建了更完整的蓝图,还获得了捕获几种菌株遗传多样性的泛基因组集合。
而现在,苹果也将成为我们的研究对象。
最近,纽约州北部的 Cornell University Orchards(康奈尔大学果园)为 Boyce Thompson Institute(博伊斯汤普森研究所)和 USDA Agricultural Research Service(美国农业部农业研究服务署)的科学家提供源材料,以构建精选的驯养和野生苹果的定相二倍体基因组组装。 https://www.nature.com/articles/s41588-020-00723-9

在 Nova Scotia,Dalhousie University(达尔豪斯大学)的研究人员正计划对来自世界上最多样化的果园之一Canada’s Apple Biodiversity Orchard(加拿大苹果生物多样性果园)的苹果进行测序,并组装一个单倍体分型的泛基因组。
“我们已经从 1,000 个苹果的果园中收集了大量性状数据。
我们的表型数据包括硬度、成熟时间和糖含量等性状,”科学家 Sophie Watts 说。
“需要改进的领域是我们的基因数据。”
凭借 2021 年 SMRT 动植物资助提供的资源,研究人员希望通过他们的研究解决几个问题。
评估在生长季节不同时间开花和成熟的苹果树上苹果的基因组可以帮助农民适应气候变化,Zoë Migicovsky 说。
“随着不可预测的天气事件的频率增加,捕获开花和成熟时间的遗传变异将成为应对气候变化影响和提高苹果恢复力的重要起点,”她补充道
HiFi 数据有助于保护苹果的生物多样性并更好地估算复杂的性状,Tom Davies 说。
“对于任何基因研究来说,高质量的参考基因组都十分珍贵,”他补充到。“使用 HiFi reads 构建的完整基因组将帮助我们精确‘放大’疑似可以控制重要性状的基因组重要区域,例如成熟时间和营养成分。” ”
马铃薯的长读长测序
苹果只是可以从基因组测序中受益的一种农产品。 您是否有想过土豆是如何生长的? 马铃薯是全球超过 13 亿人的主食,也是全球的重要作物。 不幸的是,马铃薯是一种无性繁殖的四倍体作物,由于害虫防治、贮藏和运送给农民,碳足迹很大。 它们也容易感染许多疾病,并且在历史上,没有育种遗传收益
为了解决这些问题,科学家们投入大量工作,将马铃薯重新发明为一种基于近交系的二倍体作物,通过种子繁殖。
 二倍体马铃薯是可以实现的。事实上,近 70% 的天然马铃薯种质,包括野生物种和地方品种,都是二倍体。 但科学家们在试图构建自己的二倍体杂交种时面临着许多挑战。 由于有害突变,很难开发出高度纯合的自交系,但这又是培育杂交马铃薯的先决条件。
二倍体马铃薯是可以实现的。事实上,近 70% 的天然马铃薯种质,包括野生物种和地方品种,都是二倍体。 但科学家们在试图构建自己的二倍体杂交种时面临着许多挑战。 由于有害突变,很难开发出高度纯合的自交系,但这又是培育杂交马铃薯的先决条件。
为了克服这些障碍,需要深入了解马铃薯基因组。 由 Sanwen Huang 领导的中国科学家团队转而采用 HiFi 测序,以帮助培育出一代纯正肥沃的马铃薯品系。
正如他们在《Cell》上的一篇论文中详述的那样,他们依靠基因组分析在整个育种过程中(从确定基因组纯合性和有害突变的数量,到单倍型信息)做出决策,从而推断有益和有害等位基因之间的联系。
由此产生的杂交种在温室和田间条件下均生长良好,产量高,果实和块茎富含类胡萝卜素和干物质。
这一成果意味着基因组辅助回交(一种用于种子作物的常规过程)现在可用于马铃薯育种,将马铃薯转化为更高产作物和更有营养食品具有巨大潜力。
“本研究将马铃薯育种从一种缓慢、非累积的模式转变为一种快速迭代的模式,从而为农民和消费者带来广泛优势,”作者写道。
“我们甚至预计,杂交育种和基因组设计将使马铃薯成为其最具破坏性的疾病晚疫病的非宿主,使其成为一种更环保的作物。”
用于育种的更优的牛基因组
尽管作物已经构成了一系列独特的障碍,但在尝试对牛等母-父-子家系中千兆碱基大小的哺乳动物基因组进行测序时,不同性质的挑战又接踵而至。

由于短读长测序和不完整参考基因组的固有局限性,科学家们很少在牛科领域研究较大的结构变异 (SV) 和位于重复或具有挑战性区域的变异。
一个国际研究小组最近在 bioRxiv 上发布了一篇预印本文章,他们在文中描述了牛泛基因组集合的构建。
研究人员检查了三个反映不同育种策略(品种内、亚种间和种间)的牛家系,并构建了 10 个单倍体分型的组装,这些组装被认为比当前的单倍型合并的牛科参考序列更加连续和正确。
由此产生的泛基因组为研究 SNP 和插入缺失之外的牛科系统发育提供了机会。 研究人员指出,一些结构变异无法从短或嘈杂的长读取比对中获得,例如 QRICH2 中的串联重复。 即使具有良好的长读长比对,例如插入 HSPA1B,也仍旧存在其他挑战。
但是,PacBio 长读长测序方法提供了解决方案。
“与更长但更具有高错误的 ONT reads 相比,HiFi reads 的更高精准度通常更有利于组装质量测量和提高着丝粒和端粒区域的完整性。”文中写道。
在这种情况下,准确度差异很明显。
作者观察到:“Shasta 组装在使用 ONT reads 和短读长进行一轮抛光后平均 QV 为 41.5,而 hifiasm 组装在没有任何抛光的情况下 QV 可达到 47.6”。
他们接着说:“…与 Shasta 组装相比,hifiasm 组装的基本错误减少了 4 倍,这表明 HiFi 数据能够以更少的步骤实现更高的质量。”
他们指出,基于 HiFi 的组装序列需要更少的计算和存储资源。 生产单倍体分型的 HiFi 组装需要大约 600 个 CPU 小时和 200 GB 的峰值内存使用量,而等效的基于 ONT 的 Shasta(加上抛光)组装需要 2,200 个 CPU 小时和 750 GB 的峰值内存使用量。
